イントロダクション
先日の『おまえはもうRのグラフの日本語表示に悩まない (各OS対応)』に少しだけ書いたとおり, RandomForest::MDSplot() は ggplot2 ではなく標準グラフィックスを使っている. これに言及したのはもともとR-wakalangで質問があったから. 質問者は標準グラフィックスの扱いに慣れていないようだった. 前回の記事でも ggplot2 を使えと強くお薦めしたので ggplot2 版を作成してみた1.
ちなみにRでランダムフォレストをやるなら今は ranger のほうが便利だが, 教師なしランダムフォレストは今の所用意されていない2から, とりあえず randomForest のみ対応する.
使用法
結論から言うとこういう関数を作った. 必要パッケージは ggplot2 と GGally のみ.
ggMDSplot <- function(model, label = model$y, k = 2, ...){ d <- stats::cmdscale(1 - model$proximity, k = k) d <- as.data.frame(d) colnames(d) <- paste0("Dim", 1:NCOL(d)) d$label <- label if(NCOL(d) - 1 == 1){ d$index <- 1:NROW(d) ggplot2::ggplot(d, ggplot2::aes(x = index, y = Dim1, color = label)) + ggplot2::geom_point() } else if(NCOL(d) - 1 == 2){ g <- ggplot2::ggplot(d, ggplot2::aes(x = Dim1, y = Dim2, color = label)) + ggplot2::geom_point() } else if(NCOL(d) - 1 >= 2) { g <- GGally::ggpairs(d, ggplot2::aes(color = label), columns = colnames(d)[-NCOL(d)], ...) } return(g) }
標準パッケージの引数はどうも省略しすぎでわかりづらいので変更しているが, 概ね元の MDSplot() と同じようになっている. まず model はランダムフォレストのモデルオブジェクト, label は学習データに対応するラベル. 元の関数ではなぜか必須引数だったが, そうするメリットがまったく思いつかなかったのでデフォルトでモデルオブジェクトに含まれるラベルを参照することにした (ラベルなしで学習させてもNULL値になるだけなのでエラーが発生したりはしない). よって, 最低限必要なのはランダムフォレストのモデルオブジェクトを指定することのみである.
出力は ggplot2 なので, ggplot2 の構文にそってテーマを変えたりできる. もちろん ggthemes を使うこともできる. 2次元までは ggplot2 だが, 3次元以上はオリジナルは散布図行列で表示するため, GGally を使用している. ggMDSplot() のオプション引数は全て k = 3 以上のときに呼び出される GGally::ggpairs() のオプション引数である.
よって, ggMDSplot() はオリジナルと全く同じ画像を出力するわけではない. というかそれはわざわざ ggplot2 を使う意味がない.
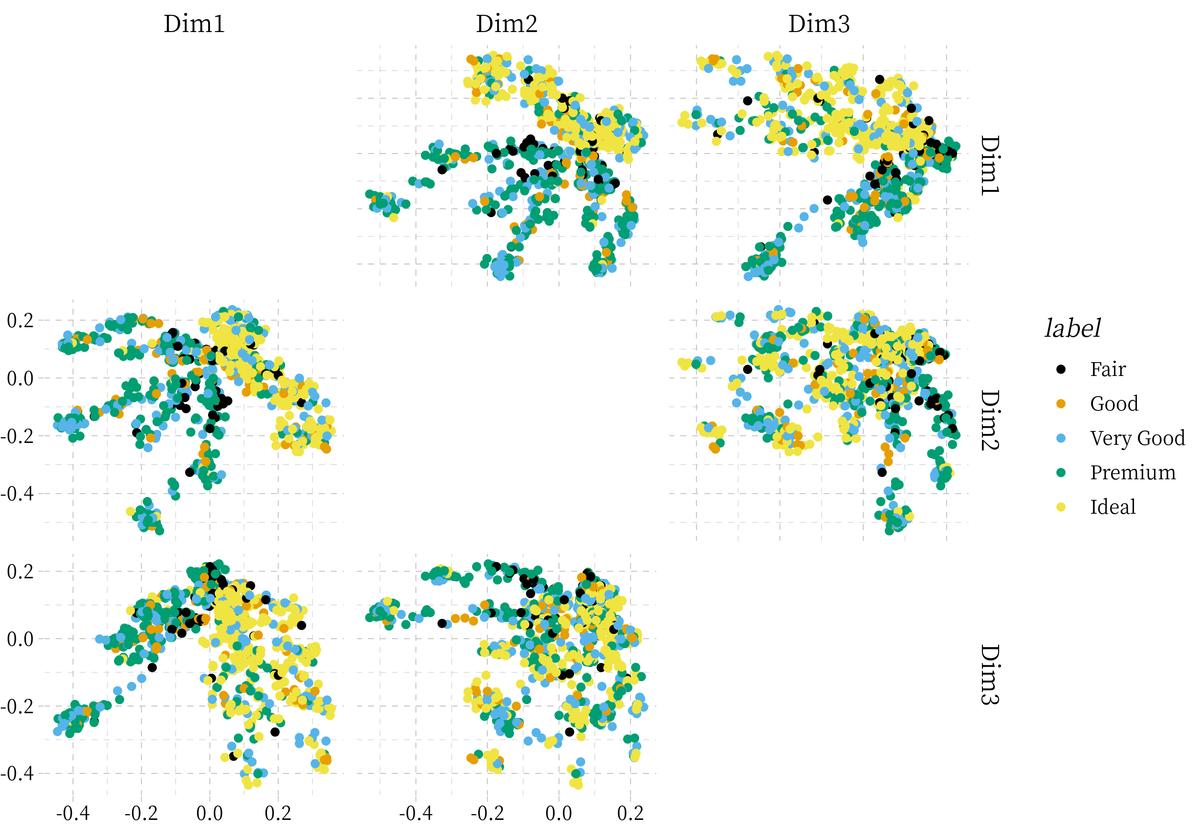
まずはオリジナルのMDSplot()とggMDSplotのデフォルトを比較する.
require(tidyverse) require(randomForest) require(GGally) require(ggthemes) data(diamonds) set.seed(42) diamonds <- diamonds[1:1000, ] rf_mds <- randomForest(cut ~ color + clarity + table, data = diamonds, proximity = T) ggMDSplot(rf_mds, k = 3) MDSplot(rf_mds, rf_mds$y, k = 3)
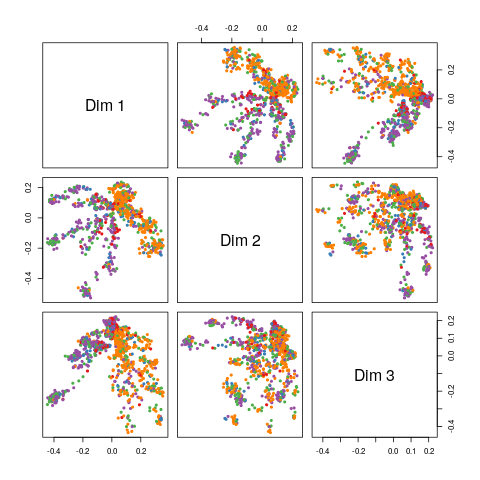
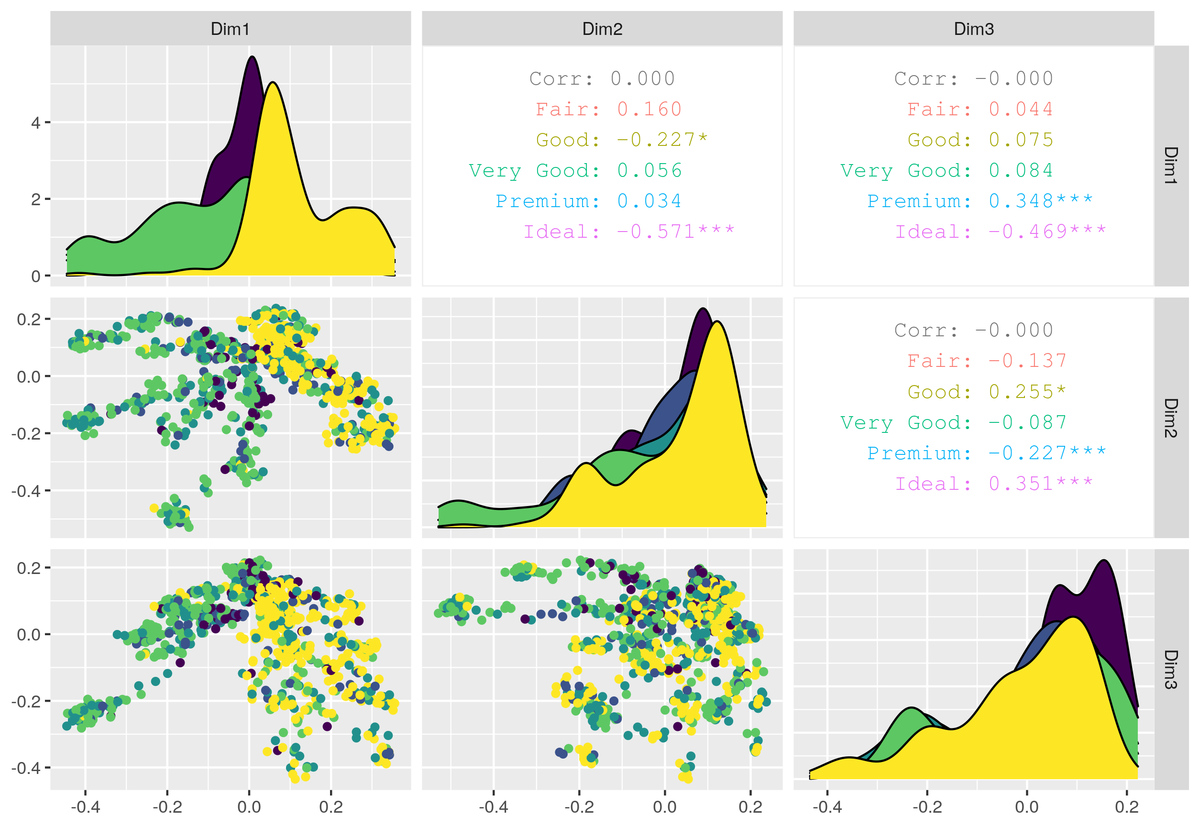
ggMDSplotだが, どうしても MDSplot() に似せたいのなら, 例えば散布図行列の対角成分を消し, 上側三角成分も散布図にするといった変更が必要になる. さらに凡例の追加と, ggthemes を使って色パレットなどを変更してみた.
ggMDSplot(rf_mds, k = 3, legend = 4, diag = list(continuous = "blankDiag"), upper = list(continuous = "points")) + theme_pander(base_family = "Noto Serif CJK JP") + scale_fill_colorblind() + scale_color_colorblind()
ggplot2と同様にカスタマイズ可能そもそも私はGGallyをあまりつかったことがない3ので, GGally の設定はよそで確認してほしい. 例えば『GGallyパッケージのggpair関数を使いこなすための覚え書き』とか
いちおう私よりも前に作った人がいるが, 完全とは言えない中途半端な状態なので改めて作り直した. https://www.slideshare.net/zgmfx20a/random-forests↩︎
作るみたいな話もあるが宙ぶらりん状態なので様子見 https://github.com/imbs-hl/ranger/issues/234↩︎
どこに何が描いてあるか, どこに注目すべきか, 見せる人間に説明しなければわからないようなグラフは描く必要がないというのが私の方針なので, そうなると雑多な散布図行列はまっさきに使用すべきグラフの候補から外れるため, この手のパッケージはめったに使わない.↩︎